AIによる解析案件があり、普段一切触ることのない、DICOMファーマットに出会いました。
DICOM
wiki引用
医療画像をAIで解析することは、全世界で活用できるだろうし、医者によって判断の違いを補助できるとても素晴らしいことだと思います。
まぁもちろん、最終的な判断はお医者様なのは当然ですが
もし、医療関係の仕事を請けることがあれば、DICOMフォーマットにも出会うことだと思うので、この記事が参考になればと思います。
この記事では下記を説明します。
- DICOMに必要なライブラリ
- データの読み込み
- 画像保存
事例
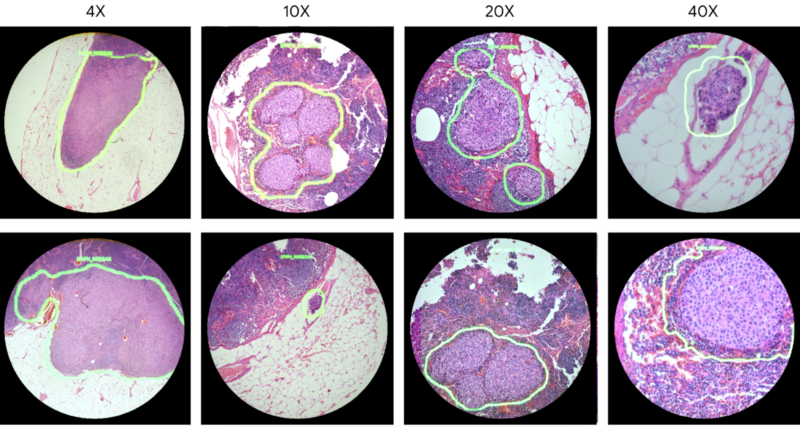
私がよくみる事例は、下記のようなガン検知AIです。
顕微鏡とARディスプレイを使っているやり方も、とても参考になります。
1年前くらいに問い合わせしたことがありますが、開発段階で、そもそもカメラ自体が
数百万するようなものらしいです。
pythonコード
ここからDICOMを読み込むコード説明になります。
ライブラリ
ライブラリには「pydicom」を使います。
インストールは下記で実行
pip install git+https://github.com/pydicom/pydicom.gitデータ読み込み
下記コードでデータが読み込めます。
import pydicom
d = pydicom.dcmread(g_drive + '/sample.dms')
# print(d)
print(d.Manufacturer)
rows = int(d.Rows)
cols = int(d.Columns)
print("Image size.......: {rows:d} x {cols:d}, {size:d} bytes".format(rows=rows, cols=cols, size=len(d.PixelData)))
#画像データ読み込み
img = d.pixel_array
img.shapeメーカー、画像サイズ、データシェイプなどをついでに確認しています。
Terumo Corporation
Image size.......: 512 x 512, 531515614 bytes
(6875, 512, 512)
データがおおきいなと思っていたら、グレースケールの画像が6875枚入っているようです。
画像表示
画像表示はここまでくれば、普通の画像と一緒です。
from matplotlib import pyplot as plt
import cv2
import numpy as np
%matplotlib inline
#1枚目を表示
plt.imshow(img[0])
plt.gray()
plt.show()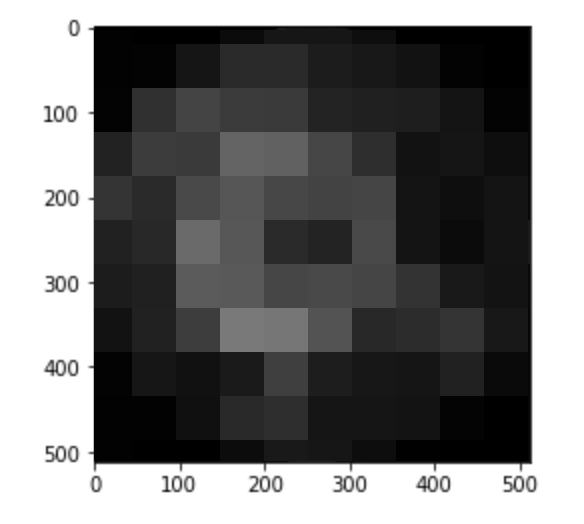
画像保存
画像保存も普通ですね。opencvを使って、下記コードで保存します。
cv2.imwrite('保存パス', img[0])
